解决方式:
近期遇到R包pRRophetic的报错解决方案,这个包是常用的通过基因表达量预测药物反应的是工具。
主要参考了微信公众号的文章
近期很多人遇到这样的问题:
>pRRopheticPredict(data, drug, selection=1)”
“Error in if (class(testExprData) != "matrix") stop("ERROR: \"testExprData\" must be a matrix.") : the condition has length > 1”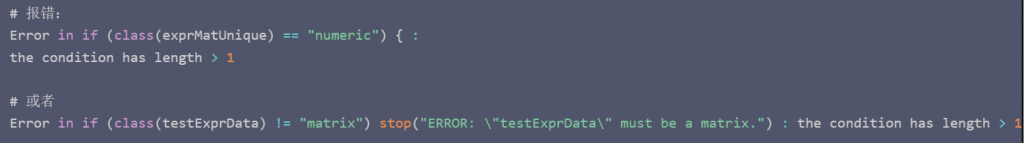
这个报错,应该是由于R语言升级后对Matrix的属性认识不同造成的。
我们看一下Matrix的帮助文档
?matrix在里面写了这样一句话:
A matrix is the special case of a two-dimensional array. Since R 4.0.0, inherits(m, "array") is true for a matrix m.
说Matrix是一种特殊的array,自从R4.0开始,就继承了array这个属性。
直白的表示就是,一个maxtrix,以前查看属性只有一个matrix,但是现在有多个,举例如下:
mdat <- matrix(c(1,2,3, 11,12,13), nrow = 2, ncol = 3, byrow = TRUE,
dimnames = list(c("row1", "row2"),
c("C.1", "C.2", "C.3")))
mdat
class(mdat)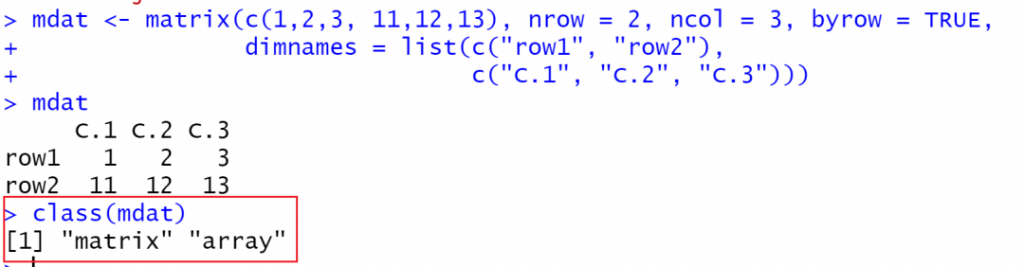
这样带来的问题是,如果某一步需要判断是不是matrix才能往下操作,之前的代码就很容易报错
比如
if(class(mdat) == "matrix"){
print("The data is wright, Go ahead!")
}他会报错

这个报错信息,与遇到的问题一样,说明找到了问题所在。只要我们把作者R包里面的源代码中涉及到矩阵判断的部分全部改一下,只提取第一个matrix就行(将原代码中的class(testExprData) != "matrix")全部修改为class(testExprData)[1] != "matrix")),
举例如下:
if(class(mdat)[1] == "matrix"){
print("The data is wright, Go ahead!")
}就是只提取第一个属性matix

另外,原r包的作者在2023.12.12修改了r包解决了此问题,但并未打包,因此需要手动修改原始的r包并重新打包,详细内容参见作者github,与微信公众号修改的方法一致(我将公众号修改的此包放到了github,按此下载)。同时建议安装r tools,以保证安装时正确编译。
参考资料
https://cloud.tencent.com/developer/article/2353766
https://mp.weixin.qq.com/s/SwnmKCjzURNV4B848nCvsw
https://www.bilibili.com/opus/908571997584228358
https://cloud.tencent.com/developer/article/1887707






Comments | NOTHING